HitGen has established a drug discovery research platform for small molecules and nucleic acid drugs centered on the design, synthesis and screening of DNA encoded chemical libraries (DELs), fragment-based drug discovery (FBDD) and structure-based drug design (SBDD) technologies.
Advantages of using DEL Selection for Protein Degrader Discovery
DEL selection is an affinity-based approach to recognize compounds interacting with the target, including the compounds modulating target function(s) or simply binding to the protein. The architecture of a DNA Encoded Compound is very similar to proteolysis targeting chimeras (PROTAC) as shown in the following figure. Both DEL compound and PROTAC molecule require covalent linkage of two molecules with known attachment points that have minimal impact to the binding.
More importantly, DEL selection is able to identify affinity binders for Protein Of Interest (POI) and E3 ligases, therefore it has advantages in creating stronger intellectual properties and exploring therapeutic benefit from novel E3 ligase(s) by their tissue distribution differences.
In a traditional DEL selection, where target is seeking for compound, the direct readout of DEL Selection is the absolute number of DNA sequences (ideally prepotential to number of compounds). In principle, when both POI and E3 ligase are used in the DEL selection, we are able to identify compounds binding to both proteins, using POI and/or E3 ligase as control(s).
POI-Compoun-E3 Ligase Ternary Complex Stability is the Key for Protein Degradation
In order to prove the above theory, we have used JQ1 as BRD4 binder, thalidomide as CRBN binder, linkers with different lengths to test whether we are able to distinguish the ternary binding stability. Specifically, six PROTAC molecules and dBET1 (a well-known BRD4 degrader, Biochem Biophys Res Commun. 2018, 497(1):410-415.) were made and their IC50 values were measured. From the IC50, we are not able to know which one(s) bind to both BRD4 and CRBN to form more stable complex.
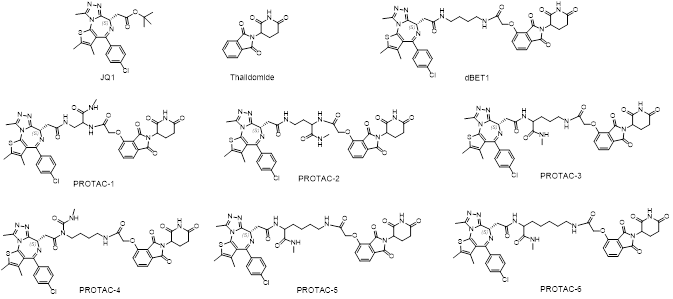

Then these 6 PROTAC molecules and dBET1 were evaluated with a FRET based ternary complex formation assay, they clearly showed different capabilities in ternary complex formation. BRD4 degradation experiment in MV4;11 confirmed the differential degradation of these 6 PROTAC molecules.

DEL Selection Approach Positively Correlates to Complex Stability
In order to confirm that DEL selection is able to distinguish complex stability, we made 6 corresponding PROTAC-DNA conjugates to see if different recovery can be observed from a DEL selection. In the “Conjugate Recovery Experiment”, Conjugate 1~6 were pooled into DEL library; a DEL selection process was conducted and the corresponding DNA sequence counts were compared (DEL selection please refer to “PROTAC DEL Selection”). Conjugate-4 is readily recognized as the optimal degrader molecule since it has significantly higher sequencing count, which is corresponding to number of compounds binding to CRBN-BRD4 complex. The length is consistent to optimal linker length in dBET1. On the other hand, recovered DNA sequences from CRBN and BRD4 samples did not give the correct trends.
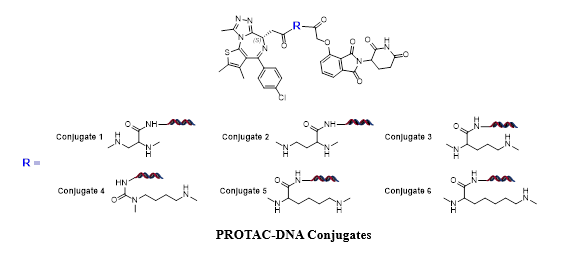
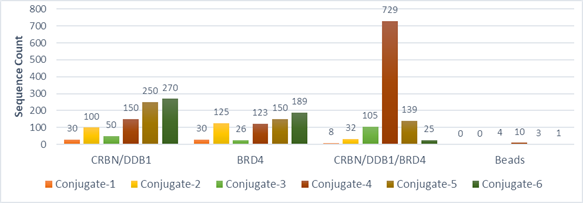
Spike-in conjugate Recovery from CRBN:BRD4 DEL Selection
Discovery of Novel BRD4 Degraders using HitGen PROTAC DELs
HitGen PROTAC DELs include E3 ligase binders and diverse chemical structures, where thalidomide is to target CRBN and R1, R2, R3 are linker and POI binding variation, respectively.
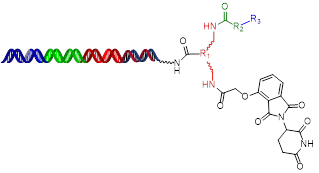
CRBN Focused Protein Degradation DEL
PROTAC DEL Selection Plan and Objectives
For this specific DEL, thalidomide has been approved clearly to bind CRBN, therefore DEL Selection against CRBN alone was not included. The Selection Plan is outlined below.

The aim is to identify any novel compounds binding to both BRD4 and CRBN over BRD4, subsequently proving that the PROTAC molecules with strong signals in Sample 2 result better BRD4 degradation.
PROTAC DEL Selection Results
For clarity, a specific plane with given R3 in the cubic presentation of DEL compound enrichment was compared between sample 1 and sample 2 (Blank sample not shown).
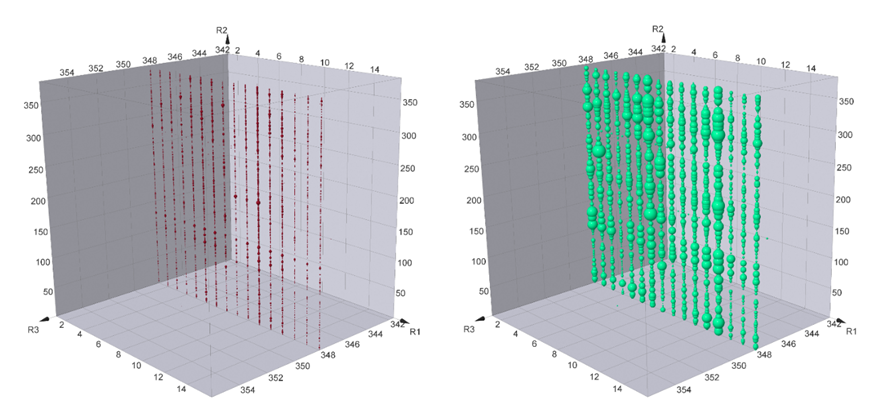
Enrichment Comparison for Sample 1 vs Sample 2. Sample 1 only use BRD4 as the protein for DEL Selection; Sample 2 use both BRD4 and CRBN for DEL Selection.
Plane R3=348 clearly suggests that many compounds on this plane are preferentially bind to both proteins (Sample 2) rather than just BRD4 (Sample 1). Meanwhile, there are also many compounds bind to both samples similarly. All the compound structures for BRD4 binding are different from JQ1 or any reported BRD4 binders. In order to compare the structure similarity, tarnimoto similarity is used to describe how different they are. When Tarnimoto similarity is greater than 0.8, two molecules are considered as similar. The representative compounds are selected and their tarnimoto similarity scores.
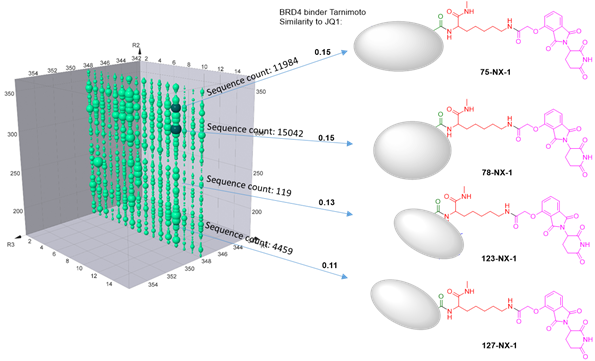
Presumably, the compounds with bigger bubbles (higher sequencing counts) form more stable complexes than that with smaller bubbles. In order to prove this, we have selected 4 representative compounds with different sequence counts in sample 2 (11984, 15042, 119, 4459) for Off-DNA synthesis and protein degradation evaluation (Western Blot). These 4 compounds are named as 75-NX-1, 78-NX-1, 123-NX-1, and 127-NX-1, respectively. Using dBET1 as a reference, compound 75-NX-1, 78-NX-1 with higher sequencing counts observed very similar BRD4 degradation in MV4;11 cell; whereas compound 123-NX-1 almost did not observe BRD4 degradation. Compound 127-NX-1 did not show BRD4 degradation either.
PROTAC compound 75-NX-1 and its BRD4 binding part (57-NX-1, structure not shown) are evaluated in anti-proliferation assay in MV4;11 (CCK-8), the results showed that compound 75-NX-1 has much higher activity than 57-NX-1 (Compound 57-NX-1 was found as a BRD4 inhibitor).
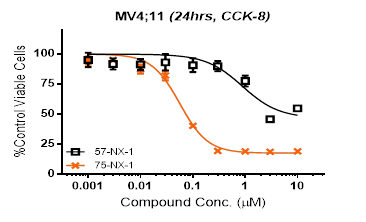
Anti-proliferation Assay for PROTAC and its Precursor
Taken the above together, it suggests that DEL selection is an effective approach to discover or optimize PROTAC compounds to find the most stable POI:Compound:E3 ligase complexes which consequently will have higher chance to achieve protein degradation in cell. We are fully aware that there are additional factors could affect the protein degradation such as PROTAC compound solubility, lipophilicity, orientation and etc, but there is no doubt that DEL selection approach provides a very effective tool for ternary complex formation (an essential step for protein degrader optimization).
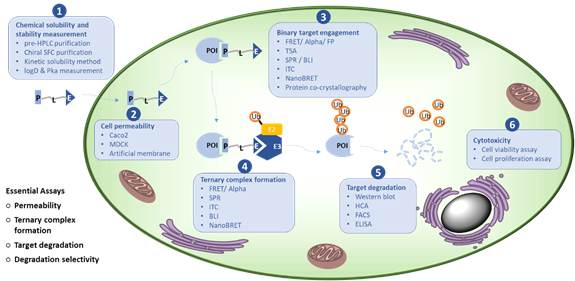
Protein Degradation Related In Vitro Assays
We use cookies to provide a better web experience.
By using our site, you acknowledge our use of cookies and please read our Cookie Notice for
More information